きゅうもるの中の人
@cuemolnohito
Followers
925
Following
5K
Media
704
Statuses
13K
東京科学大・難治研/生体分子の立体構造とシミュレーション・深層学習/メンバー募集中(https://t.co/NSrR73gTGQ)/CueMolという生体高分子の構造可視化ソフトウェアを作っています.
東京都
Joined February 2011
1週間だけの公開らしいので、興味のある方は是非ご笑覧ください・・・(TVでうまく話すのって中々難しいですね)
加藤英明教授がBSテレ東の「今からサイエンス」に出演されました。放送は11/13から1週間、番組HPより視聴可能となります。 https://t.co/uMWl9iFTpw
1
21
145
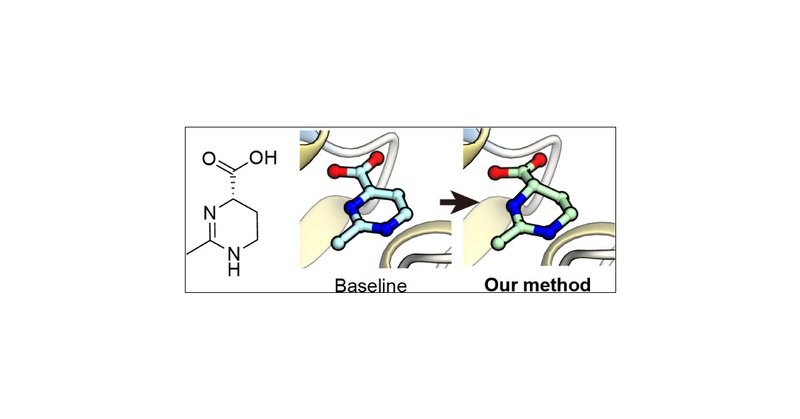
先日掲載された論文、Improving Stereochemical Limitations in Protein–Ligand Complex Structure Predictionの解説ブログを書きました!
cddlab.io
AlphaFold3やその派生モデルBoltz-1によるタンパク質-リガンド複合体構造予測において、 リガンドの立体化学(キラリティ、結合長、結合角)の再現性に問題があることが知られています。 本研究では、この問題を解決するための新しい手法「拘束誘導推論(restraint-guided inference)」を開発し、 キラリティの再現率を100%に到達させることに成功しました。...
1
11
48
The Ishitani (A02) and Terada (A02) groups developed an improved protein–ligand complex structure prediction based on Boltz-1. This software perfectly reproduces the chirality specified in the input chemical structure. Available at: https://t.co/DSDaOSYvF6
https://t.co/DVPcMCLOpt
pubs.acs.org
AlphaFold3 has revolutionized biology by enabling the prediction of protein complexes with various biomolecules, including small molecular ligands. However, the ligand structures predicted by the...
1
15
41
AF3(といっても実際はBoltzですが)の立体構造予測の化合物の立体化学を改善する論文()が、ACS Omegaに掲載されました! https://t.co/xElKOHSBas
pubs.acs.org
AlphaFold3 has revolutionized biology by enabling the prediction of protein complexes with various biomolecules, including small molecular ligands. However, the ligand structures predicted by the...
0
9
52
研究室のページにブログを作ってみました。ぼちぼち更新していければと思います。 https://t.co/XtniKjvFDO
cddlab.io
こんにちは。計算創薬科学研究室(CDD Lab)の石谷です。 この度、研究室のウェブサイトにブログを開設することにしました。 このブログでは、研究室での日々の活動や研究の話題、学会参加報告など、様々な情報を発信していきたいと思います。
0
8
21
Predicting protein complexes in biosynthetic gene clusters https://t.co/6zqwv3gFcX
#biorxiv_bioinfo
biorxiv.org
Biosynthetic gene clusters (BGCs) are contiguous genomic regions that encode diverse, non-homologous proteins required for the production of specific natural products. Their genetic diversity...
0
7
27
プレプリントを公開しました
Predicting protein complexes in biosynthetic gene clusters https://t.co/6zqwv3gFcX
#biorxiv_bioinfo
1
19
87
@Ag_smith
@MasaSomiya
@akg_entrance
@AiNiitsu212 + 分子研・小杉さんらとともに、タンパク質デザインに関する研究会を企画しています!ぜひご参加ください! (定員60名予定) 【日時】2025/12/10 (水) 13–17時 【場所】東京科学大学 湯島キャンパス (対面のみ) 【参加登録】 https://t.co/olXSAOh55P
1
21
39
Multi-objective optimization for designing structurally similar proteins with diverse sequences 1. A new method combining ProteinMPNN and the multi-objective optimization algorithm NSGA-II has been proposed to design proteins that are structurally similar to a target protein
2
14
84
井上さん達が発見したChR024について、特に吸収波長とイオン輸送に着目して構造機能相関を調べたpreprintが公開されました。024がBCCR/PLCRクレードのChRよりポンプ型ロドプシンと似ていた点は意外でした。後電気生理実験を全て自ラボで行った初論文という点でも感慨深いです https://t.co/6tmxUH5zeR
新たに発見された長波長吸収型チャネルロドプシン(ChR)に関する研究のPreprintが公開されました。今回の分子は名古屋大学・竹内一郎先生のグループと長年開発してきた機械学習予測をもとにして発見されました。また、従来のChRと進化的に独立していることも興味深い点です🧬 https://t.co/Y0rbPp978H
3
12
79
今日刊行の実験医学 増刊 「構造生命科学 AlphaFold時代にどう活かす?」に、PFNの武本さん、富田さんらとともに蛋白質・化合物の相互作用予測について書かせてもらいました。 https://t.co/pXLlkuFTTS
yodosha.co.jp
あらゆる生命科学研究者が構造データを活用できる時代が到来!AlphaFoldを含む最先端の構造予測AI,クライオEMやクライオETで構造データを取得し,タンパク質の機能解析・デザイン・創薬にどう活かすのか,豊富な事例とともに研究戦略を解説.
0
9
25
8月26日発売の科学雑誌ニュートン最新号で、Topic AI創薬 最新レポートを監修しました 試し読みページはこちら https://t.co/qolUk0daVF
newtonpress.co.jp
最新号(2025年12月号 )の特集は「AI時代の哲学 」です。
2
18
47