Soya Shinkai
@ShinkaiSoya
Followers
160
Following
729
Media
0
Statuses
121
Physical biology and bioinformatics of the dynamic 3D genome. Senior Scientist @BDR_RIKEN
Kobe, JAPAN
Joined May 2022
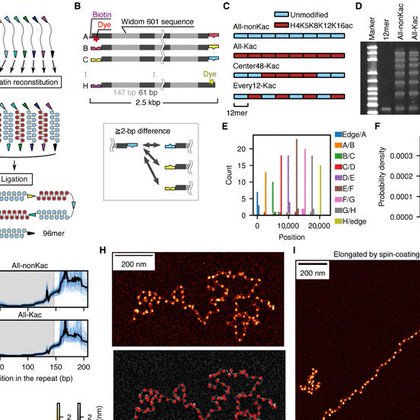
遺伝子スケールのクロマチン物理を研究するため、ヒストン修飾を制御した96-mer (20 kb) の再構成系を確立しました。1分子観察・原子間力顕微鏡・in vitro Hi-Cにより特定の修飾パターンが構造や動態をどう変えるか調べています。論文のビジュ結構いいのでぜひ見てください!
science.org
Reconstituting 20-kb chromatin shows that tuning acetylation alone reshapes its folding, dynamics, and contact domain formation.
2
24
100
We are currently recruiting a tenure-track Assistant Professor for our laboratory. We would be grateful if you could review the application details at the following link and consider the opportunity. https://t.co/W5kCZVHEEf
1
7
7
バイオイメージングデータの共有と再利用のためのプラットフォーム「SSBD」@ssbd_ja の新たな紹介動画を公開しました。SSBD:repositoryには学術誌上で発表された全てのバイオイメージング関連データをどなたでも論文発表に合わせて公開することが可能です。 https://t.co/jF5H4mX4EV
0
14
23
今月の生物物理誌(Vol. 65 No. 3) @bpsjp に寄稿させていただきました。2023年の大規模Hi-C解析について紹介しています。村山さんと同じ号です(・∀・) よろしければ御覧ください。 https://t.co/ADNJrYljOq
0
7
13
🧬My retrospective chromatin review is out@Proc Jpn Acad: https://t.co/0te20pvDUj Textbook models of 30-nm fibers are fading. A new paradigm: chromatin forms liquid-like domains—locally dynamic, yet globally stable at chromosome level (viscoelastic)—supporting genome function.✨
1
21
95
GloBIAS #BioimageAnalysis #Conference 2025 – we have opened the registration for our 2025 in-person event. Please check the webpage https://t.co/pUr17U2a80 for more information and the link to the registration form.
0
19
23
We’ve released PHi-C2 v2.1.0, a major update from v2.0.13! • Now supports .hic format as input • Allows exclusion of NaN-rich regions using a --tolerance threshold • Enhanced preprocessing pipeline Install via: pip install phic 🔗 [ https://t.co/9gEeKIdXDd]
github.com
PHi-C2 allows for a physical interpretation of a Hi-C contact matrix - soyashinkai/PHi-C2
0
3
12
Huge congratulations to @katsu_s_minami, @kazu_maeshima, and all collaborators on the publication of this fantastic paper!
Our new paper is out @ScienceAdvances👇 https://t.co/VF4XGubD3B 🧬Our Repli-Histo labeling marks nucleosomes in euchromatin and heterochromatin in live human cells. 🔍@katsu_s_minami et al.have developed a chromatin behavior atlas within the nucleus. 1/2
0
0
9
I am seeking an Assistant Professor in my lab. If you are interested, please contact me! 📩 ゲノムダイナミクス研究室で助教を公募中です。ご興味のある方はぜひご連絡ください! RTお願いします。 🔄 日本語版👇👇👇
0
36
53
実験医学での連載をベースにした生物画像解析の本が3/24に発売されます!初歩的な画像解析を既にやっていて、さらに深く発展させたい人をターゲットにしているのと、スクリプトを書く入門的な内容にもなっていて、必要な人の助けになればと思います。 https://t.co/4huzP5Ydbp
yodosha.co.jp
画像解析スキルをさらに高めたい方に! あなたの研究目的にあった画像解析法をデザインするための基本戦略とツールの種類・使い方を「型」で体得する超・実践型教本.コーディング未経験でも,最先端の機械学習・深層学習ツールを取り入れた解析自動化や,バイアスの少ない解析を実現できる力が身に付く.
2
36
169
おめでとうございます🎉
SMC複合体を介したアーキアゲノムの構造化機構についての論文がNat. Commun.誌に掲載されました! This work has been published in Nature Communications! The manuscript has been significantly improved thanks to the reviewers' valuable comments! https://t.co/JBaqgjWg35
1
0
0
今週末! 2025年2月16日(日)at 新代田FEVER MASS OF THE FERMENTING DREGS presents 「デイドリームなんかじゃない vol.17」 O/S 12:00/12:30 前売 ¥3500+1D¥600 学生割引 ¥2500+1D¥600 高校生以下 無料+1D¥600 *割引には学生証提示必須 🎫 https://t.co/KT7erEhE4m
0
24
106
九州大学の落合さんと大石さんとの共同研究が出版されました。エンハンサー−プロモーター相互作用が機能するための時空間制御の仕組みの一端に、一歩近づくことができました。これからもPHi-Cを活用して、さらに面白い展開を目指していきたいと思います。
science.org
Active state during transcriptional bursting associates with proximities between genomic regions and protein accumulation.
1
8
81
Thank you so much, @Hiro_Ochiai_En and @OhishiHiroaki, from the very beginning of this study. Discovering the importance of viscosity around the active gene through a new usage of our PHi-C simulations was truly exciting!
🚨 Exciting news! 🚨 Our latest paper is now published in Science Advances! 🎉 "Transcription-coupled changes in genomic region proximities during transcriptional bursting" 👉 Read it here:
0
0
8
コロナ禍隔てて、あっという間の第4回Hi-C研究会、大盛況です。 オーガナイザーの岡部さんの尽力とともに、中戸さんが編集した本の労いもあり、楽しい1日です
0
5
16
Thanks for organising the great meeting. Please add @Hiro_Ochiai_En
This was a very dense and fun meeting! Thanks to all the speakers! @jmuiuc @a_z_usa__ @IchiroHiratani @kazu_maeshima @ShinkaiSoya (as far as I could find...
1
0
1
I reminded Zwanzig's solvable model for GLE derivation [J Stat Phys (1973)] and incorporated its projection method. Personally, this application of Zwanzig’s projection formalism was my highlight.
0
0
0
Although previous studies had derived a GLE for the Rouse model, they employed a "specialized" approach based on transformations to the normal coordinates.
1
0
0
In this study, we derived that the motion of an individual bead in the linear elastic network model—a polymer model used in our genome modeling approach, "PHi-C"—can be rigorously described by a Generalized Langevin Equation (GLE).
1
1
0